AWS MarketplaceでDragenを使用してゲノムをアライメントする
目的
Dragenの性能、料金を実際に使ってみて確認する。
やること
AWS Marketplaceを使ったことがないので使い方を学ぶ。
どのように料金が発生->消滅するのか確認する。
実行日
2018年7月
AWS DRAGENのインストールガイドがあります。このガイドに簡単にDragenを使用できます。このブログはインストールガイドにしたがってDragenをインストールしていき、わからないことや突っかかったことをまとめた
AWS Marketplace製品の起動方法はAWS コンソールからインスタンス起動時に、その製品を選べばよいとのことです。
AWS Marketplace を使用した製品のサブスクライブ
Amazon EC2 起動ウィザード: AMI を検索し、ウィザードからインスタンスを直接起動できます。詳細については、「AWS Marketplace インスタンスの起動」を参照してください。
インスタンス起動->AWS Marketplace->"Dragen"検索で出てきました。
使用可能な3つのリージョンからオレゴンリージョンを選択しました。

Free TrielでDRAGENが1日タダで使えるようです。
価格の内訳がありました。
**Pricing Breakdown:
F1.2xlarge: $10.35 (DRAGEN) + $1.65 (Compute) = $12.00/hr
Per Genome: $12.00hr/0.8 genomes = $15
Per Exome: $12.00hr/5.45 exomes = $2
F1.16xlarge: $22.10 (DRAGEN) + $13.20 (Compute) = $35.30/hr
Per Genome: $35.30hr/1.76 genomes = $20
Per Exome: $35.30hr/12 exomes = $3
Marketplaceを使用する権限がないためエラーになりました。
User: arn:aws:iam::xxxxxxxxxx:user/kchiba-ci is not authorized to perform: aws-marketplace:ViewSubscriptions on resource: *
AWS Marketplace Full Accessを付与してエラーを解消しました。
AWS Marketplace: AWS Marketplace : Help
メモ:1
I started a free trial for a product I purchased on AWS Marketplace but never used the software during the trial period. Can I restart my trial since I didn’t get any use out of it?
試用期間を確認しながらAMIを使用すれば、FreeTrialを超過しないように気をつけられそうです。
メモ:2
You can view your usage in the billing console. Billing will be recorded under the AWS Marketplace section: https://console.aws.amazon.com/billing/home#/
Marketplaceの請求額はコンソールをみて確認すればよいとのことです。簡単です。
とりあえず動かしてみる

こちらをcontinueします。

使用するインスタンスを選択します。
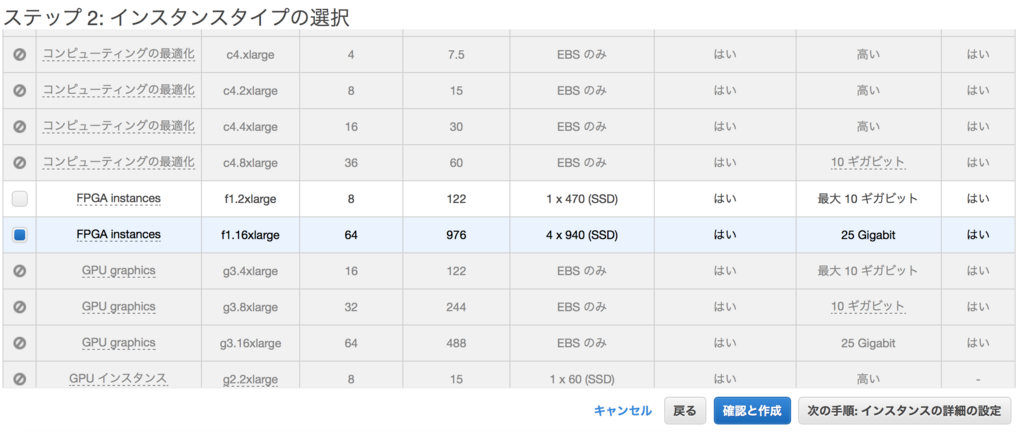
S3からデータをストリーミングでインプットできるようにロールの設定をします。
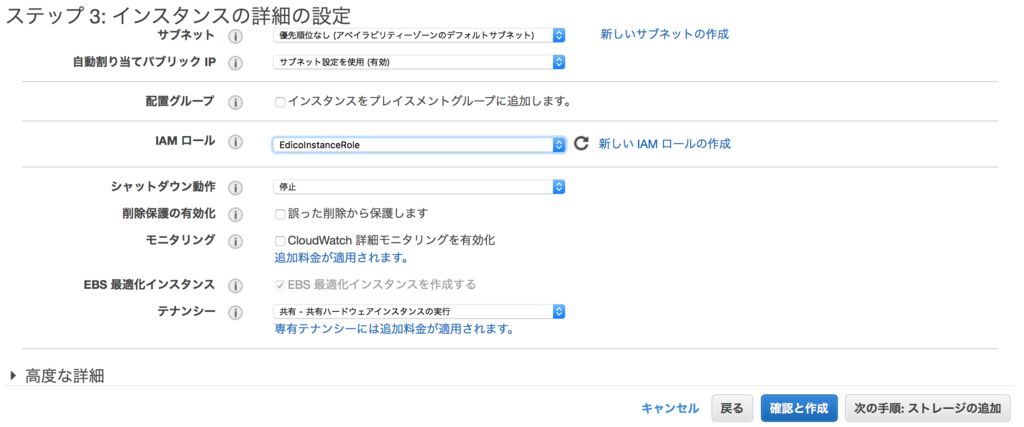
結果のBAMファイルとかはephemeralに置かれる。940GiBx4サイズがあれば十分。

タグづけは任意とのことなので名前だけつけておく

画面を開くとセキュリティーグループは新規が選択されていて、名前までデフォルトで入力済み。このままセキュリティーグループを作成する。

警告はでてる(いつもの)。キーペアを使うので安全とする。
作成ボタン押下後にキーを設定する。

キーペアを指定する画面がでてくる(いつもの)
既存のキーペアを指定して作成。
作成中の画面です。

起動時間は通常のEC2を立ち上げるのと同じくらいすぐだけど、2/2チェックが15分くらいかかった。
ssh ログイン後 早速、不穏なログが出力される。

立ち上げたらやること
pip install awscli --upgrade --user
・AWS Configure
リファレンスゲノムの設定。
とりあえずFASTAファイルをダウンロードする。
[centos@ip-172-31-18-26 GRCh37]$ pwd
/ephemeral/GRCh37
[centos@ip-172-31-18-26 GRCh37]$ ls -l GRCh37.fa
-rw-rw-r-- 1 centos centos 3189752728 7月 6 03:58 GRCh37.fa
リファレンスゲノムのHashTableの作成
[centos@ip-172-31-18-26 GRCh37]$ time dragen --ht-reference GRCh37.fa --output-directory /ephemeral/GRCh37 --build-hash-table true
作成完了 13分くらいかかった
DRAGEN finished normally
real 13m18.982s
user 83m34.607s
sys 3m40.037s
リファレンスゲノムのHashTableの作成 RNA version
[centos@ip-172-31-18-26 GRCh37_RNA]$ time dragen --ht-reference GRCh37.fa --output-directory /ephemeral/GRCh37_RNA --build-hash-table true --ht-build-rna-hashtable true
作成完了 22分
DRAGEN finished normally
real 22m5.017s
user 128m6.781s
sys 6m34.592s
AWSのEC2 GPUインスタンスでNanopore Guppyを実行する
目的
- Nanopore GuppyをGPUで実行する
EC2の環境
- p3.2xlarge
- ubuntu 16.04
手順
- GPUが使用出来るEC2インスタンスを起動する
- AWS EC2のLinux インスタンスに NVIDIA ドライバをインストールする
- Nanopore Guppyのインストール
- Nanopore Guppyを実行する
1. GPUが使用出来るEC2インスタンスを起動する
AWS EC2 の GPUの製品シリーズ(2019/07/24現在)。
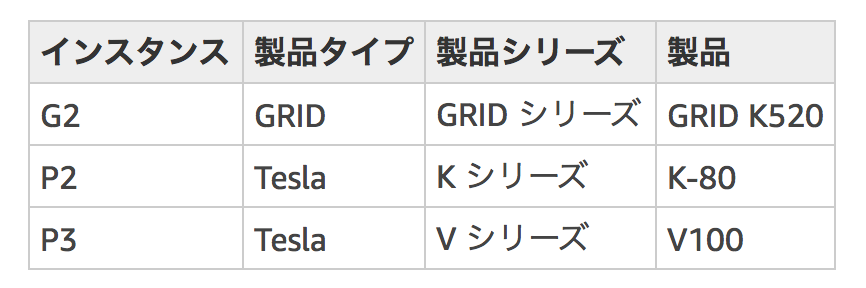
Nanoporeのウェブページをみると、製作者がTesla V100 を使っているようだ。Guppyと互換性があるだろうと考えてTesla V100にする。

GPUつきのEC2インスタンスは、値段高い。(仕方ないとは思うけど率直な感想)
コンソール画面からp3.2xlargeのインスタンスを起動。テスト用なので小さいインスタンスを選択した。EC2インスタンスを立てる処理に特別なことはしていないので説明は省略。
2. AWS EC2のLinux インスタンスに NVIDIA ドライバをインストールする
http://www.nvidia.com/Download/Find.aspx からNvidia Driverをダウンロードする。
EC2インスタンス環境に合わせて、Tesla V100、ubuntu16.04、CUDA Toolkit 最新バージョンを選択して「SEARCH」ボタンを押下
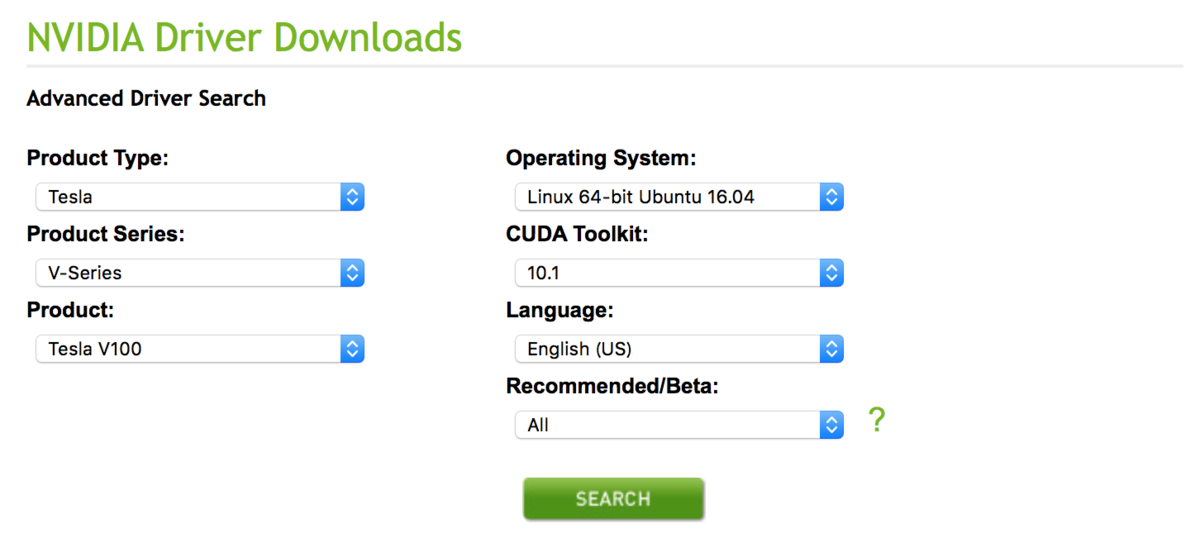
2つのドライバ候補が表示されたので、新しいバージョンのversion 418.67の 「Tesla Driver for Ubunbu 16.04」をクリック(リンクになってる)

Dowonload画面に遷移した。この後にEC2インスタンス上からwgetしてドライバをダウンロードするので「DOWNLOAD」ボタンのURLをコピーしておく。
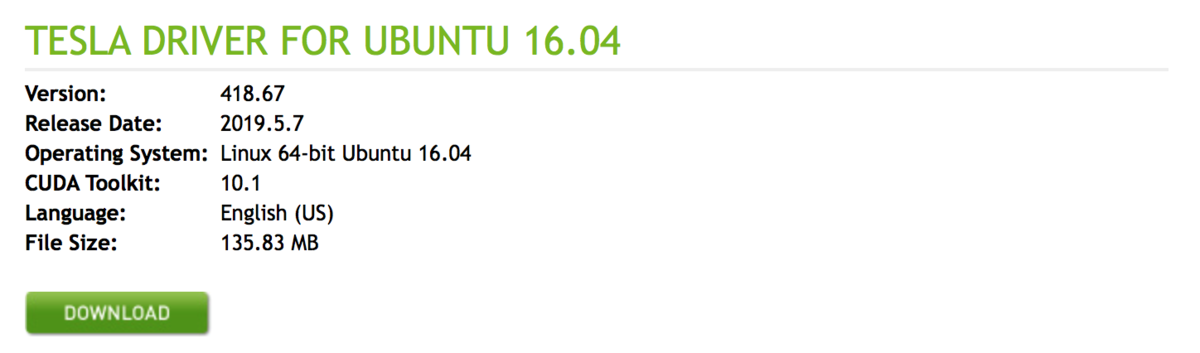
EC2インスタンスにログインして、NVIDIAドライバをインストールする。基本的に以下URLのアマゾンの公式ドキュメントの通りに実行する
https://docs.aws.amazon.com/ja_jp/AWSEC2/latest/UserGuide/install-nvidia-driver.html
EC2インスタンスに接続
# パッケージの更新
$ sudo apt-get update -y
# パッケージのアップグレード
$ sudo apt-get upgrade -y linux-aws
# インスタンス再起動
$ sudo reboot
EC2インスタンスに再接続
# gccとカーネルヘッダーパッケージをインストール
$ sudo apt-get install -y gcc make linux-headers-$(uname -r)
# NVIDIA グラフィックカード用のnouveauオープンソースを無効化する
$cat << EOF | sudo tee --append /etc/modprobe.d/blacklist.conf
blacklist vga16fb
blacklist nouveau
blacklist rivafb
blacklist nvidiafb
blacklist rivatv
EOF
# 以下の行を追加する
$ sudo vi /etc/default/grub
GRUB_CMDLINE_LINUX="rdblacklist=nouveau"
# grubの再構築
$ sudo update-grub
# NVIDIA ドライバのダウンロード。NVIDIAのホームページから検索したドライバのURLを指定する。
$ wget https://www.nvidia.com/content/DriverDownload-March2009/confirmation.php?url=/tesla/xxx.xxx/nvidia-diag-driver-local-repo-ubuntu1604-xxx.xxx.amd64.deb&lang=us&type=Tesla
# NVIDIA ドライバーのインストール
$ sudo /bin/sh ./NVIDIA-Linux-x86_64-xxx.xxx.run
# インスタンスの再起動
$ sudo reboot
EC2インスタンスに再接続
# NVIDIA ドライバがインストールされたか確認する
$ nvidia-smi -q | head
3. Nanopore Guppyのダウンロード
NanoporeのサイトからGuppyをダウンロードする。NanoporeのユーザのみGuppyのダウンロードが可能。
# Guppyのダウンロード $ wget https://xxxxx/xxxxx/ont-guppy_3.1.5_linux64.tar.gz # 解凍する $ tar xzvf ont-guppy_3.1.5_linux64.tar.gz
4. Nanopore Guppyを実行する
コマンドのオプションを確認する
guppy_basecaller
--input 当研究室でNanopore PromethIONでシークエンスしたデータの一部を入力する
--flowcell シークエンス時に使用したFlowcell
--kit シークエンス時に使用したKit
--save_path 出力ディレクトリ
-x auto GPUを使用するときにこのオプションを使用する
--cpu_threads_per_caller ベースコールあたりのCPUスレッドの数。GPUを使用しないときにこのオプションを指定する。
GPUモード
実行時間 114秒
ubuntu@ip-xxx-xx-xx-xxx:~/tools$ ont-guppy/bin/guppy_basecaller --flowcell FLO-PRO002 --kit SQK-LSK109 --input ~/guppy/input --save_path ~/guppy/output_gpu -x auto
ONT Guppy basecalling software version 3.1.5+781ed57
config file: /home/ubuntu/tools/ont-guppy/data/dna_r9.4.1_450bps_hac_prom.cfg
model file: /home/ubuntu/tools/ont-guppy/data/template_r9.4.1_450bps_hac_prom.jsn
input path: /home/ubuntu/guppy/input
save path: /home/ubuntu/guppy/output_gpu
chunk size: 1000
chunks per runner: 1000
records per file: 4000
num basecallers: 4
gpu device: auto
kernel path:
runners per device: 2
Found 5 fast5 files to process.
Init time: 4247 ms
0% 10 20 30 40 50 60 70 80 90 100%
|----|----|----|----|----|----|----|----|----|----|
***************************************************
Caller time: 114628 ms, Samples called: 1694657572, samples/s: 1.4784e+07
Finishing up any open output files.
Basecalling completed successfully.
CPUモード
実行開始から2時間で、全体の10%くらいの進捗(遅いorz
ubuntu@ip-xxx-xx-xx-xxx:~/tools$ ont-guppy/bin/guppy_basecaller --flowcell FLO-PRO002 --kit SQK-LSK109 --input ~/guppy/input --save_path ~/guppy/output_cpu --cpu_threads_per_caller 2
ONT Guppy basecalling software version 3.1.5+781ed57
config file: /home/ubuntu/tools/ont-guppy/data/dna_r9.4.1_450bps_hac_prom.cfg
model file: /home/ubuntu/tools/ont-guppy/data/template_r9.4.1_450bps_hac_prom.jsn
input path: /home/ubuntu/guppy/input
save path: /home/ubuntu/guppy/output_cpu
chunk size: 1000
chunks per runner: 1000
records per file: 4000
num basecallers: 4
cpu mode: ON
threads per caller: 2
Found 5 fast5 files to process.
Init time: 2344 ms
0% 10 20 30 40 50 60 70 80 90 100%
|----|----|----|----|----|----|----|----|----|----|
*******
EC2インスタンスの値段が高いのでCtrl+Cして処理を中断しました。
GPUモードでFASTQファイルが正常にできた。
Minimap2やngmlrを使用して作ったFASTQのアライメントをしていこうと思う。
メモ
- Albacoreの実行も試みたが、自分が持っているシークエンスデータのフローセルとキットの組み合わせ(--flowcell FLO-PRO002 --kit SQK-LSK109)が存在しなかった。新しくシークエンスしたFAST5データはguppyを使えということだろうと解釈した。
AWS EFSのファイル読み込みのスループットを計測する
AWS EFS Oregon regionのファイル読み込みのスループットを計測する。複数のEC2から同時にファイルを読み込んだ場合のスループットも計測する。
また、すべてのEFSは100 MiB/秒のスループットまでバーストするとマニュアルに記載があるが、読み込み時にバーストされているか確認する。
方法
1. EFSを立ち上げる。5GBのファイルをEFSに配置。
2. EC2 を立ち上げる。5GBのファイルをEFS⇨EC2へコピーする。
3. コピーにかかった時間を計測する。転送速度を算出する。
マシンスペック
EFS
Performance mode Max I/O,
Encrypted No,
Metered size 105GB,
Availability zone us-west-2a
EC2
AMI ubuntu-xenial-16.04-amd64-server-20180522 (ami-db710fa3),
Instance type m4.large,
volume tpye gp2 (8GB),
Availability zone us-west-2a
・マウントにはEFS Mountを使用した。
・Metered sizeが大きいほどスループットのバースト時間は長くなる。そのためEFSにファイルを>100GB 配置している。
Burst to 100 MiB/s for up to 72 minutes each day, or
Drive up to 5 MiB/s continuously
英文は下のサイト(マニュアル)から引用
https://docs.aws.amazon.com/efs/latest/ug/performance.html
・EC2がスループットのボトルネックにならないようなスペックにしている。
結果
| EC2の数 | ave(real time) | スループット (EC2 1台あたり) | スループット * EC2の数 |
|---|---|---|---|
| 1 | 93.51 | 53.47MiB/s | 53.47MiB/s |
| 2 | 101.95 | 49.04MiB/s | 98.08MiB/s |
| 4 | 203.33 | 25.49MiB/s | 101.96MiB/s |
| 8 | 407.73 | 12.26MiB/s | 98.08MiB/s |
・EFSは100MiB/sの回線を共有して使用する。(バースト時)
・EC2 1台だと50MiB/sくらい。100MiB/sの速度はでない。
・スループットはave(real time)から算出しているため、実際のスループットとは数MiBのズレがある。
マニュアルをみてもファイルシステムごとに100 MiB/sのスループットが使用できるという認識で間違いないと思う。
All file systems, regardless of size, can burst to 100 MiB/s of throughput, and those over 1 TiB large can burst to 100 MiB/s per TiB of data stored in the file system. For example, a 10 TiB file system can burst to 1,000 MiB/s of throughput (10 TiB x 100 MiB/s/TiB).
英文は下のサイト(マニュアル)から引用
https://docs.aws.amazon.com/efs/latest/ug/performance.html
結果詳細
EC2 1台
| # | real time | user time | sys time |
|---|---|---|---|
| 1 | 93.51 | 0.01 | 5.26 |
EC2 2台で同時読み込み
| # | real time | user time | sys time |
|---|---|---|---|
| 1 | 101.76 | 0.00 | 4.69 |
| 2 | 102.14 | 0.00 | 4.85 |
EC2 4台で同時読み込み
| # | real time | user time | sys time |
|---|---|---|---|
| 1 | 202.66 | 0.00 | 4.96 |
| 2 | 203.27 | 0.00 | 4.81 |
| 3 | 203.62 | 0.00 | 4.89 |
| 4 | 203.77 | 0.02 | 4.98 |
EC2 8台で同時読み込み
| # | real time | user time | sys time |
|---|---|---|---|
| 1 | 405.71 | 0.00 | 5.08 |
| 2 | 406.76 | 0.00 | 4.91 |
| 3 | 407.62 | 0.00 | 4.90 |
| 4 | 407.72 | 0.02 | 5.05 |
| 5 | 407.89 | 0.00 | 4.95 |
| 6 | 408.54 | 0.02 | 5.30 |
| 7 | 408.74 | 0.02 | 5.15 |
| 8 | 408.91 | 0.00 | 5.10 |
バーストクレジットについて
Cloud WatchのBurstCreditBalanceとPermittedThroughputを使用すると理解しやすい。
BurstCreditBalance
・データコピーを実施したらバーストクレジットが減っていった。
・データコピーをしていないときはバーストクレジットが回復していく。Metered size が10GBより100GBの方が回復量が大きい。
PermittedThroughput
・EFSの最大スループットが表示される。今回はクレジットに余裕があったため、105MB/sから下がることはなかった。
メモ
・EFSにファイルを配置したからといって、即座にMetered sizeが増えるわけではない。どこかのタイミングでディスク使用量が増える。
・EFSへの大量のアクセスが予想されたため、Max I/Oを選択したが、マニュアルの「Using The Right Performance Mode」を実施しGeneral Purpose Performance Modeが使用可能であるかチェックするべきであった。
https://docs.aws.amazon.com/efs/latest/ug/performance.html
・EFSでは英語版のマニュアルが日本語版よりかなり充実している。
Azure Batch サンプルプログラムのカスタマイズ
目的
Azure Batchのpythonサンプルプログラムを、ゲノムシークエンスデータ解析に使いやすいようにカスタマイズしたのでメモしておく。
カスタマイズしたプログラム
オリジナルのサンプルプログラム
azurebatchmonの実行環境
Azure 仮想マシン Standard DS1 v2 (1 vcpu、3.5 GB メモリ) Ubuntu
Azureのアカウントが必要
・Azure アカウント (メインのアカウント)
・Azure Batchアカウント( Azure Portal で Batch アカウントを作成する | Microsoft Docs )
・ストレージアカウント( Azure Portal でストレージ アカウントを作成… | Microsoft Docs )
Pythonのモジュールのインストール
# azure-batchとazure-strageにアクセスするためのpythonパッケージ
pip install azure-batch
pip install azure-storage
Azurebatchmon概要
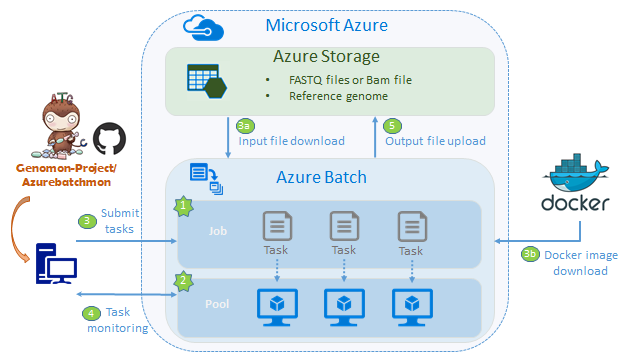
事前準備
リファレンスゲノム、FASTQ Fileと実行ScriptをAzureストレージにアップロードしておく。Docker imageをDockerHubに登録しておく
1.AzurebatchのJobを作成
2.Azurebatchのpoolを作成
3.タスク(実行するプログラム)の登録
Azureストレージから解析に必要なデータをダウンロード
docker imageをpullしてくる
解析処理の実行
4.タスクをモニタリングする
5.解析が完了したら、結果をAzure Storageにアップロードする
Azure クイック スタート - Batch ジョブの実行 - Python | Microsoft Docs
カスタマイズのポイント
1.シークエンスデータは数十~数百GBのビックデータなのでローカルからのアップロードはしない
ゲノムデータはストレージに置いておく。Microsoft Genomicsもデータはストレージはじまりでストレージ終わり。このやり方が無難。
2.ストレージのデータに対するダウンロードとアップロードにazcopyを使う。
azcopyの利点
・recursiveが可能
・Storageアカウント、コンテナ名とファイル名を1つのpathとして記載できるので便利。(cliだとファイル名とは別にコンテナ名を指定する必要があった)
・destに指定したファイルパスに、ディレクトリがない場合は作成される
サンプルコード(ダウンロード
azcopy \
--source https://storage_account.file.core.windows.net/my_container/ \
--destination /mnt \
--source-key storage_account_key \
--recursive
サンプルプログラムにあるTaskAddParameterを用いてストレージからのファイルダウンロードは、recursiveができないっぽいので使わなかった。
3.Pool作成時のStartタスクでDockerをインストールする。
そしてタスクで解析ツールが入っているDocker imageをダウンロードして、解析処理を実行する。
Pool作成時のStartタスクでDockerをインストール
pool_start_commands = [dockerインストール]
PoolAddParameter(command_line=common.helpers.wrap_commands_in_shell('linux',pool_task_commands),
・・略・・)
タスクでDockerを使った解析処理を実行する
commands = [docker run イメージ]
tasks.append(batch.models.TaskAddParameter(
'azmon-task-{}'.format(idx),
common.helpers.wrap_commands_in_shell('linux', commands),
user_identity=run_elevated))
便利だったところ
・スタートタスクが失敗してもリトライされる。
・VMが起動してタスクが投入されるところをコンソールから確認できる。
困ったところ
・pool中のVMのタスクが失敗した場合の戻り値をどうやってとるかわからない。
・東日本リージョン混んでる(2018年3月時点)low priority VMを使ったけど、かなり割り込みが多かった。→西日本リージョンに移動した。
改善ポイント
・オートスケーリング機能を使用して、idleのVMをdeleteした方が良い。
・タスクにリトライ機能があるので使った方が良い。
・ログをストレージに保存するように設定する。現時点ではプールの削除とともにログも消える。
クラウドって
・データのリージョン間コピーのお値段高いので気をつける。(Azureに限らないけど)
最後に
改善ポイントはまだあるけど、ほかにもやりたいことあるし、3月にできるだけやろうと考えていたので、Time's upとしよう。
Salt StackでGoogle Cloud Platform (GCP) をマネージメントしたい
E
目的
インフラ構成管理ソフトについて勉強中。AWSで使ってみて、使用感が良かったので、GCPでも使用してみる。
この記事でやること
SaltStackの公式ドキュメント [Getting Started With Google Compute Engine] をやってみる。
Salt StackでGCPをマネージメントするために用意しておくこと
- なし ←事前準備が"なし"というのがGCPのすごいところ!
Salt Stackを実行するCompute Engineを用意する
次のCompute Engineをコンソールから作成した。
ssh-keyを作成して、Compute Engineのメタデータに登録する
一般的なやり方なのでわかりやすい。以下のサイトを見ると簡単に登録できる
SSH 認証鍵ペアによるインスタンス アクセスの管理 | Compute Engine ドキュメント | Google Cloud Platform
Salt Stackのインストール
作成したCompute Engineにログインし、SaltStackの公式ドキュメント [Ubuntu] を参照してインストールを行う。このsalt-masterをインストールした環境が管理サーバとなる。
# apt-getのアップデー
apt-get update
#一般的な ネットワークアクセスを行うためのパッケージ
apt-get install salt-api
# public cloud VM をマネージメントするパッケージ
apt-get install salt-cloud
# 管理サーバに入れるパッケージ
apt-get install salt-master
# クライアントサーバに入れるパッケージ
apt-get install salt-minion
# リモート操作をするためのパッケージ
apt-get install salt-ssh
# 分散リモート実行システムパッケージ
apt-get install salt-syndic
# おまじない
salt-cloud -u#おまじないについて
# file_ignore_glob関連のWARNINGが大量に出力されてしまうので、対応しておく
公式ドキュメントに書いておいてほしいですね。
Create a Service Account
サービスアカウントのキーを作成してダウンロードする。下記のマニュアルをみて、デフォルトのサービスアカウントのjsonファイルを生成して、Compute Engineにアップロードしておく
サービス アカウント キーの作成と管理 | Cloud Identity and Access Management のドキュメント | Google Cloud Platform
Salt Cloudを使用してクライアントサーバを構築する
2つのCompute Engine構築のための設定ファイルを用意する。これによりインフラ構築環境を再現することができる。
1つめ:/etc/salt/cloud.providers
# プロバイダー名。設定をこの名前を使って取り込む重要な名前
gce-config
# Set up the Project name and Service Account authorization
project: "your-project-id"
service_account_email_address: "123-a5gt@developer.gserviceaccount.com"
service_account_private_key: "/path/to/your/NEW.pem"
# masterのprivate ip アドレスをいれておく
minion:
master: xx.xx.xx.xx
# このままでいい
grains:
node_type: broker
release: 1.0.1
driver: gce2つめ:/etc/salt/cloud.profiles
# プロファイル名。salt-cloud実行時にこの名前を使って指定する。
my-gce-profile:
image: ubuntu-1604
size: n1-standard-1
location: asia-east1-c
network: default
subnetwork: default
use_persistent_disk: True
delete_boot_pd: False
deploy: True
make_master: False
provider: gce-config
ssh_username: your_login_user
ssh_keyfile: /home/your_login_user/.ssh/id_rsa
↑で使用しているssh_keyfileのpublic key (id_rsa.pub) をCompute Engineのメタデータに登録する。ほかに本家のマニュアルを見ればもっと細かいことをやっているが、こちらは最小設定。
Salt Cloudを実行する
# Usage: salt-cloud -p <プロファイル名> <新たに作られるインスタンスのName(任意)>
# インスタンスを立てる
salt-cloud -p my-gce-profile kchiba-test-instance
MasterからMinionのコマンドを起動する
# minionサーバのコマンドが実行できた。
# salt kchiba-test-instance test.ping
kchiba-test-instance
: True
# pythonも実行できる
# salt kchiba-test-instance cmd.run 'python --version'
kchiba-test-instance:
Python 2.7.12
便利っぽい。
おかたづけ(インスタンスを削除する)
#Compute Engineをdestroyするコマンドの実行 # salt-cloud -d kchiba-test-instance The following virtual machines are set to be destroyed: gce-config: gce: kchiba-test-instance Proceed? [N/y] y# Masterの設定も初期化されたかをみてみる。 # salt-key Accepted Keys: # kchiba-test-instanceが設定されていたのが消えている
Compute Engineを削除すると同時にsalt-masterとsalt-minionの関係も解消してくれた。
使ってみて感想
Compute Engineインスタンスを簡単に作成できる。
ssh-keyなどGoogle独自の方法ではなく、一般的な方法が使えるのでわかりやすい
ほとんどAWSと同じコマンドだった。
おわり。
Azure BatchでDNAシークエンスデータをアライメントしてみる
目的
Azure BatchでDNAシークエンスデータをアライメントしてみる。勉強目的。
この記事でやること
Azure Batchでbwaを実行してみる。以下の記事に沿って勉強する。
チュートリアル - Python 向け Azure Batch SDK を使用する | Microsoft Docs
インストール方法やAzure Batchを実行するためのscriptは↑このページを参照した。今回はAzure BatchでBWAを実行することに主眼を置く。
実行環境
Azure 仮想マシン Standard DS1 v2 (1 vcpu、3.5 GB メモリ) Ubuntu
アカウントが必要
・Azure アカウント (メインのアカウント)
・Azure Batchアカウント( Azure Portal で Batch アカウントを作成する | Microsoft Docs )
・ストレージアカウント( Azure Portal でストレージ アカウントを作成… | Microsoft Docs )
Pythonのモジュールのインストール
# azure-batchとazure-strageにアクセスするためのpythonパッケージ
pip install azure-batch
pip install azure-storage
Azure Batchってなに?
・並列処理が簡単にできる
・コンピューティングリソースを自動的に拡大/縮小できる
解析したい検体数の仮想マシンが自動的に立ち上がることを期待したい!
ゲノム解析でとても効果的な機能
・低優先度VMが使用できる。VMの割り込みが起こったタスクは再度キューに登録される。
・コンピューティングノードの最大数が指定できる。
プールとは
・VMをデプロイする領域
・VMのスケール(最低→最大VM数)の指定をする
・AZ_BATCH_NODE_SHARED_DIRとあるが、物理的にshareのなディスクにおいているわけではない
ジョブとは
・タスクの置き場、タスクをキューで登録できる
・タスクとは引数ありの実行コマンドとほぼおなじ
処理概要
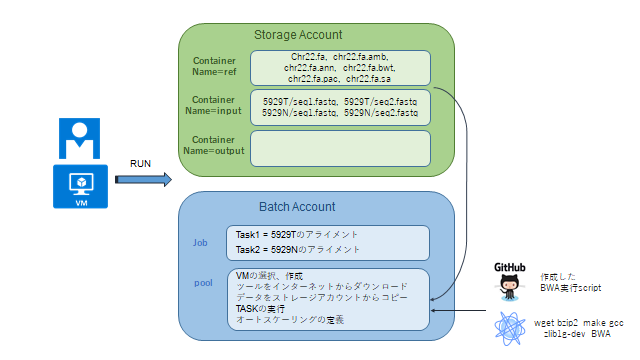
-事前準備
・リファレンスゲノム(bwa index付き)をストレージにアップロードしておく
・アライメントするFASTQファイルをストレージにアップロードしておく
・実行Scriptをgithubにアップロードしておく
-RUNすると
1. python moduleのインポート
2. Container outputを作成する
3. poolを作成する
4. Jobを作成する
5. taskを登録する→poolに空きがあればTASKが実行される
6. taskの完了を待つ
-taskに登録されるscript
・自作したpython_bwa_task.pyについて
1. python moduleのインポート
# azure-batchとazure-strageにアクセスするためのpythonパッケージ
import azure.storage.blob as azureblob
import azure.batch.batch_service_client as batch
import azure.batch.batch_auth as batchauth
import azure.batch.models as batchmodels
2. container output を作成する
# まずはblob clientを生成する。Containerの作成や削除、Blobの操作など、
# Strage Accountでの操作を行うためのインスタンス。
blob_client = azureblob.BlockBlobService(
account_name=_YOUR_STORAGE_ACCOUNT_NAME,
account_key=_YOUR_STORAGE_ACCOUNT_KEY)
# container outputの作成
output_container_name = 'output'
blob_client.create_container(output_container_name, fail_on_exist=False)
3. poolを作成する
# まずはbatch clientを生成する。poolの作成や削除、jobの作成や削除など、
# Batch Accountでの操作を行うためのインスタンス。
credentials = batchauth.SharedKeyCredentials(
_YOUR_BATCH_ACCOUNT_NAME, _YOUR_BATCH_ACCOUNT_KEY)
batch_client = batch.BatchServiceClient(
credentials, base_url=_BATCH_ACCOUNT_URL)
VMインスタンスが立ち上がった時に実行されるコマンドの作成
task_commands = [
'curl -fSsL https://bootstrap.pypa.io/get-pip.py | python',
'pip install azure-storage==0.32.0',
'apt-get update && apt-get install -y wget bzip2 make gcc zlib1g-dev',
'wget http://sourceforge.net/projects/bio-bwa/files/bwa-0.7.15.tar.bz2',
'tar xjvf bwa-0.7.15.tar.bz2',
'cd bwa-0.7.15',
'make',
'cd ..',
'cp -rp bwa-0.7.15 $AZ_BATCH_NODE_SHARED_DIR',
'wget https://github.com/ken0-1n/AzureBatchTutorialBwa/archive/v0.1.0.tar.gz',
'tar xzvf v0.1.0.tar.gz',
'cp -p AzureBatchTutorialBwa-0.1.0/{} $AZ_BATCH_NODE_SHARED_DIR'.format(_TUTORIAL_TASK_FILE)
]
# VMに指定するイメージの取得
sku_to_use, image_ref_to_use = \
common.helpers.select_latest_verified_vm_image_with_node_agent_sku(
batch_service_client, 'Canonical', 'UbuntuServer', '16')
# ユーザ権限の設定
user = batchmodels.AutoUserSpecification(
scope=batchmodels.AutoUserScope.pool,
elevation_level=batchmodels.ElevationLevel.admin)
# poolの定義, pool IDにはアカウント内でユニークな名前をつけること
new_pool = batch.models.PoolAddParameter(
id=pool_id,
virtual_machine_configuration=batchmodels.VirtualMachineConfiguration(
image_reference=image_ref_to_use,
node_agent_sku_id=sku_to_use),
vm_size=_POOL_VM_SIZE, # 'BASIC_A2'を指定
target_dedicated_nodes=_POOL_NODE_COUNT, # 2を指定。pool内に作られるVMの最大数
start_task=batch.models.StartTask(
command_line=common.helpers.wrap_commands_in_shell('linux',
task_commands),
user_identity=batchmodels.UserIdentity(auto_user=user),
wait_for_success=True))
batch_service_client.pool.add(new_pool)
4. jobを作成する
# poolは先ほど作成したpoolを指定する。
job = batch.models.JobAddParameter(_JOB_ID,
batch.models.PoolInformation(pool_id=POOL_ID))
batch_service_client.job.add(job)
5. taskを登録する
# taskを登録するときにStrageにあるBlobの情報を指定し、
ResourceFileインスタンスを生成しなければならない。そのためのメソッド。
def get_resource(block_blob_client, container_name, blob_name):
# containerにアクセスするためのtokenの生成
sas_token = block_blob_client.generate_blob_shared_access_signature(
container_name,
blob_name,
permission=azureblob.BlobPermissions.READ,
expiry=datetime.datetime.utcnow() + datetime.timedelta(hours=2))
# blobのurlの生成
sas_url = block_blob_client.make_blob_url(container_name,
blob_name,
sas_token=sas_token)
# blobの情報を設定したインスタンスの生成
return batchmodels.ResourceFile(file_path=blob_name, blob_source=sas_url)
# StrageにおいてあるFASTQファイルの情報が設定されたインスタンスを生成する
input_container_name = 'fastq'
input_files1 = [get_resourcefile(blob_client, ref_container_name, ref_file_path)
for ref_file_path in ['5929T/sequence1.fastq', '5929N/sequence1.fastq']]
input_files2 = [get_resourcefile(blob_client, ref_container_name, ref_file_path)
for ref_file_path in ['5929T/sequence2.fastq', '5929N/sequence2.fastq']]
# Strageにおいてあるリファレンスゲノムの情報が設定されたインスタンスを生成する
ref_container_name = 'ref'
ref_files = [get_resourcefile(blob_client, ref_container_name, ref_file_path)
for ref_file_path in ['hg19/chr22.fa', 'hg19/chr22.fa.amb',
'hg19/chr22.fa.ann','hg19/chr22.fa.bwt','hg19/chr22.fa.pac','hg19/chr22.fa.sa']]
# container outputにアクセスするためのtokenの生成
output_container_sas_token = get_container_sas_token(
blob_client,
output_container_name,
azureblob.BlobPermissions.WRITE)
# 実行コマンドをTaskに登録する(5929T)
command = ['python $AZ_BATCH_NODE_SHARED_DIR/python_bwa_task.py --storageaccount chiba1sa --storagecontainer output --sastoken "xxxxxxxxxx" --bwapath $AZ_BATCH_NODE_SHARED_DIR/bwa-0.7.15/bwa --refgenome hg19/chr22.fa --samplename 5929T --fastq1 5929T/sequence1.fastq --fastq2 5929T/sequence2.fastq]
tasks.append(batch.models.TaskAddParameter(
'topNtask1',
common.helpers.wrap_commands_in_shell('linux', command),
resource_files=[5929T/sequence1.fastq, 5929T/sequence2.fastq])
# 実行コマンドをTaskに登録する(5929N)
command = ['python $AZ_BATCH_NODE_SHARED_DIR/python_bwa_task.py --storageaccount chiba1sa --storagecontainer output --sastoken "xxxxxxxxxx" --bwapath $AZ_BATCH_NODE_SHARED_DIR/bwa-0.7.15/bwa --refgenome hg19/chr22.fa --samplename 5929N --fastq1 5929N/sequence1.fastq --fastq2 5929N/sequence2.fastq]
tasks.append(batch.models.TaskAddParameter(
'topNtask2',
common.helpers.wrap_commands_in_shell('linux', command),
resource_files=[5929N/sequence1.fastq, 5929N/sequence2.fastq])
batch_service_client.task.add_collection(job_id, tasks)
6. taskの完了を待つ
timeout_expiration = datetime.datetime.now() + timeout
while datetime.datetime.now() < timeout_expiration:
tasks = batch_service_client.task.list(job_id)
incomplete_tasks = [task for task in tasks if task.state != batchmodels.TaskState.completed]
if not incomplete_tasks:
return True
else:
time.sleep(1)
自作したpython_bwa_task.pyの中身
# モジュールのimport
from __future__ import print_function
import argparse
import collections
import os
import string
import subprocess
import sys
import azure.storage.blob as azureblob
if __name__ == '__main__':
# 引数の処理
parser = argparse.ArgumentParser()
parser.add_argument('--storageaccount')
parser.add_argument('--storagecontainer')
parser.add_argument('--sastoken')
parser.add_argument('--bwapath', required=True)
parser.add_argument('--fastq1', required=True)
parser.add_argument('--fastq2', required=True)
parser.add_argument('--refgenome', required=True)
parser.add_argument('--samplename', required=True)
args = parser.parse_args()
bwa = os.path.realpath(args.bwapath)
fastq1 = os.path.realpath(args.fastq1)
fastq2 = os.path.realpath(args.fastq2)
ref_fa = os.path.realpath(args.refgenome)
samplename = args.samplename
output_sam = samplename +'.sam'
error_log = samplename +'.error.log'
# bwaを実行する
sam = open(output_sam, 'w')
error = open(error_log, 'w')
proc = subprocess.Popen([bwa, 'mem', ref_fa, fastq1, fastq2], stdout=subprocess.PIPE, stderr=subprocess.PIPE)
output = proc.stdout
for line in output:
sam.write(line)
output = proc.stderr
for line in output:
error.write(line)
sam.close()
error.close()
# blobにアクセスするためのインスタンスを生成
blob_client = azureblob.BlockBlobService(account_name=args.storageaccount,
sas_token=args.sastoken)
# 結果をアップロードする
output_sam_path = os.path.realpath(output_sam)
blob_client.create_blob_from_path(args.storagecontainer,
output_sam,
output_sam_path)
error_log_path = os.path.realpath(error_log)
blob_client.create_blob_from_path(args.storagecontainer,
error_log,
error_log_path)
使ってみて感想
Pythonで記載されたAzure Batchを動かすサンプルがあったので便利。
1検体につき、1VMインスタンスが割り当ててほしいが、Auto Scaleがそのように、割り当ててくれるか検証が必要。
Docker Swarmを使用できるとのことなので、やってみなくてはならない
参考
Celery + AWS Redis で使ってみる
目的
Celeryなるツールを使ってみる。勉強目的。
この記事でやること
Celery + Redisを使ってみる。Djangoは使わない。とりあえずCeleryのマニュアルのFirst Stepsは理解したい。
First Steps with Celery — Celery 4.1.0 documentation
環境
AWS EC2 Oregon
ubuntu-xenial-16.04-amd64-server-20170619.1 (ami-835b4efa)
Celeryってなに?
・非同期処理が作れる
・スケジューリングもできる
・上記の機能を使用する際にpythonでスタンダードなツール
Message Brokerなるものが必要
タスクメッセージをCeleryに登録したり、そのタスクを取り出すBrokerなるツールが必要で、以下のツールをサポートしている。
Brokers — Celery 4.1.0 documentation
この記事ではRedisを使用してみる。Amazon SQSも気になるけど単体で使用するツールではなさそうなので、単体でも使用できそうなRedisを指定する。
Redisってなに?
以下のSlide Shareを見てもらうと手っ取り早い。RDBのデータをキャッシュする感じで使うのですかね。便利そうですね。
AWSにRedisでも立ててみる
料金について。無料利用枠があるとのことなのでcache.t2.microを使う。
AWS コンソールから作成してみる
クライアントのEC2と同じセキュリティーグループにいれて、inboudを許可したらコネクトできました。
ElastiCacheのredisをEC2で使う - Qiita
CeleryとRedisはどうやってつながるの?
Celery instanceが必要だというので立ててみる。
tasks.pyをつくる
from celery import Celery
app = Celery('tasks', broker='redis://ubuntu@your.primary.endpoint.cache.amazonaws.com')
@app.task
def add(x, y):
return x + y
Celeryデーモンの起動
celery -A tasks worker --loglevel=infoなんかサーバたった。
別のプロセスを立ち上げでタスクを投げてみる
>>> from tasks import add
>>> result = add.delay(4, 4)
<AsyncResult: bf6d2799-2461-4b42-8ee1-f5bddb867f05>
>>> result.get()
(省略)
NotImplementedError: No result backend is configured.returnされたけど見えない。(ここでは見えなくて正解。backendの処理が必要)
結果を保持する
tasks.pyを更新する。赤字の設定を追加
backendに指定したストアに結果が保持される保持する。
from celery import Celery
app = Celery('tasks', broker='redis://ubuntu@your.primary.endpoint.cache.amazonaws.com', backend='redis://ubuntu@your.primary.endpoint.cache.amazonaws.com')
@app.task
def add(x, y):
return x + y
再実行
Ctrl + C (サーバ止める)
celery -A tasks worker --loglevel=info
結果
>>> from tasks import add
>>> result = add.delay(4, 4)
>>> result.get()
8
Redisのデータがどうなっているか気になるので見てみる
redis-cliを使用する
# redisに接続
redis-cli -h your.primary.endpoint.cache.amazonaws.com
# key検索
> keys *
"celery-task-meta-xxxxxxxx-xxxx-xxxx-xxxxxxxxx"
# 登録したタスクがいっぱい出てくる
# 登録したタスクの検索
get celery-task-meta-xxxxxxxx-xxxx-xxxx-xxxxxxxxx
# 格納されてた
"{\"status\": \"SUCCESS\", \"traceback\": null, \"result\": 8, \"task_id\": \"xxxxxxxx-xxxx-xxxx-xxxxxxxxx\", \"children\": []}"
使ってみて感想
簡単に非同期処理ができてとても便利。いろいろと使えそう。
参考
http://docs.celeryproject.org/en/latest/index.html
https://www.slideshare.net/yujiotani16/redis-76504393